


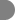

| General Information: |
|
| Name(s) found: |
gi|151940981
[NCBI NR]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae YJM789 |
| Length: | 775 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSFDRPEIYS APVLQGESPN DDDNTEIIKS FKNFILEFRL DSQFIYRDQL RNNILVKNYS 60
61 LTVNMEHLIG YNEDIYKKLS DEPSDIIPLF ETAITQVAKR ISILSRAQSA NNNDKDPENT 120
121 SMDTDSLLLN SLPTFQLILN SNANQIPLRD LDSEHVSKIV RLSGIIISTS VLSSRATYLS 180
181 IMCRNCRHTT SITINNFNSI TGNTVSLPRS CLSTIESESS MANESNIGDE STKKNCGPDP 240
241 YIIIHESSKF IDQQFLKLQE IPELVPVGEM PRNLTMTCDR YLTNKVIPGT RVTIVGIYSI 300
301 YNSKNGAGSG RSGGGNGGSG VAIRTPYIKI LGIQSDVETS SIWNSVTMFT EEEEEEFLQL 360
361 SRNPKLYEIL TNSIAPSIFG NEDIKKAIVC LLMGGSKKIL PDGMRLRGDI NVLLLGDPGT 420
421 AKSQLLKFVE KVSPIAVYTS GKGSSAAGLT ASVQRDPMTR EFYLEGGAMV LADGGVVCID 480
481 EFDKMRDEDR VAIHEAMEQQ TISIAKAGIT TVLNSRTSVL AAANPIYGRY DDLKSPGDNI 540
541 DFQTTILSRF DMIFIVKDDH NEERDISIAN HVINIHTGNA NAMQNQQEEN GSEISIEKMK 600
601 RYITYCRLKC APRLSPQAAE KLSSNFVTIR KQLLINELES TERSSIPITI RQLEAIIRIT 660
661 ESLAKLELSP IAQERHVDEA IRLFQASTMD AASQDPIGGL NQASGTSLSE IRRFEQELKR 720
721 RLPIGWSTSY QTLRREFVDT HRFSQLALDK ALYALEKHET IQLRHQGQNI YRSGV |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..122] | 2.095995 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [123..373] | 8.823909 | MCM2/3/5 family No confident structure predictions are available. | |
| 3 | View Details | [374..606] | 44.522879 | ATPase subunit of magnesium chelatase, BchI | |
| 4 | View Details | [607..775] | 44.522879 | ATPase subunit of magnesium chelatase, BchI |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)